Chapter 5.1 : Découpage par blocs
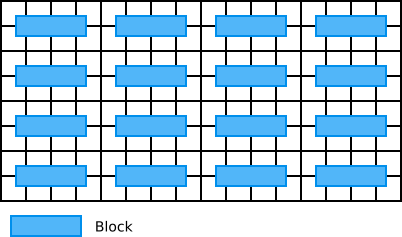
Pour réduire la quantité de mémoire utilisée à un instant donné pour traiter une image est de la découper en blocs. La figure 9 illustre le découpage simple par blocs.
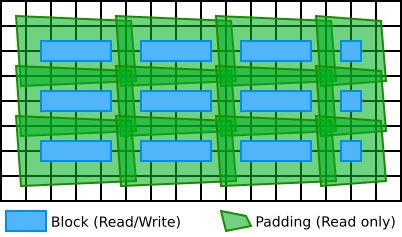
Nous allons utiliser des blocs avec un pourtour de une cellule, comme illustré dans la figure 10, ce qui permet de prendre en compte le voisinage d'un bloc.
Dans notre problématique, il est important qu'un bloc complet (avec son voisinage) tienne completement en RAM pour tous les tableaux que nous devons utiliser :
- Input concentration de l'espèce U
- Input concentration de l'espèce V
- Output concentration de l'espèce U
- Output concentration de l'espèce V
Ces
 tableaux doivent donc tenir dans le Cache L1 de taille
tableaux doivent donc tenir dans le Cache L1 de taille 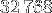 octets. Chaque tableau représente un quart des données, soit
octets. Chaque tableau représente un quart des données, soit 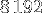 octets ce qui représente
octets ce qui représente 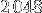 float.
float.Cela implique que les blocs doivent contenir au maximum
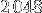 valeurs.
On pourrait, par exemple, utiliser des blocs de
valeurs.
On pourrait, par exemple, utiliser des blocs de 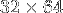 .
.Cependant, cela risque d'être un peu juste car nous devons prendre en compte le fait que nous avons besoin d'autres valeurs comme les différents coefficients de la matrice de gradien ou les différentes valeurs de diffusion, de réaction des différentes espèces.
Mais, quelle est la bonne dimension à réduire ?
Encore une fois, cela dépend de ce que l'on veut faire. Avec notre méthode vectorisée completement en fonctions intrinsèques (voir section 4.3), nous avons vu que nous pouvions réduire au maximum le nombre de valeurs de padding si le nombre de lignes est un multiple de la taille des registres vectoriels (voir figure 6). Dans ce cas, et comme notre nombre de lignes est déjà un multiple de la taille des registres vectoriels noteEt dans ce cas, c'est un multiple de la taille de n'importe quel registre vectoriel sur toutes les architectures, donc nous avons de la chance., donc nous devons réduire le nombre de colonnes.
Afin de bien cerner le problème, nous ferons une implémentation générique qui nous permettra de jouer sur la taille des blocs pour comparer les performances obtenues.